El desarrollo de las terapias biológicas ha supuesto un gran avance en el manejo de la artritis reumatoide (AR) al haber demostrado efectividad en el control de la clínica y daño radiológico. Sin embargo, entre un 20-40% de los pacientes no van a responder a estas terapias, lo que determina un alto coste económico a la vez que los expone a posibles efectos adversos, por lo que se precisa de la identificación de factores predictores de respuesta a ellos. Estos se revisan en el actual trabajo en función de su evidencia científica y se clasifican en genéticos y no genéticos. A pesar de su extensa búsqueda, en la actualidad no disponemos de potentes predictores que puedan ser utilizados en la práctica clínica diaria. Posiblemente a día de hoy sólo los factores séricos, positividad del factor reumatoide (FR) y anticuerpos antipéptido citrulinado (anti-CCP), permiten predecir la respuesta a determinados biológicos. En un futuro, probablemente gracias a las nuevas tecnologías basadas en la genómica, transcriptómica y proteómica se identificarán predictores genéticos que permita seleccionar pacientes idóneos para una determinada terapia biológica.
The advent of biological therapies has revolutionized the management of rheumatoid arthritis, demonstrating effectiveness in controlling clinical and radiological damage. However, 20 to 40% of the patients will not respond to these therapies, which are associated to a very high cost. In addition, non-responder patients are exposed to possible adverse effects. For these reasons, we need to identify predictors of response to these treatments. These predictors are reviewed in this evidence-based paper and classified into genetic and non-genetic. Despite extensive search, nowadays there are no predictors powerful enough to be used in regular clinical practice. Serum factors, the presence of rheumatoid factor and anti-cyclic citrullinated peptide antibodies, are the only factors currently being used to predict the response to specific biological therapy. In the future, probably thanks to new technologies based on genomics, transcriptomics and proteomics, it will be possible to identify genetic predictors of response to biological drugs that will allow us to select suitable patients for a specific biological therapy.
La artritis reumatoide (AR) es una enfermedad inflamatoria sistémica crónica, con predisposición genética, de etiología desconocida caracterizada por una sinovitis simétrica erosiva.
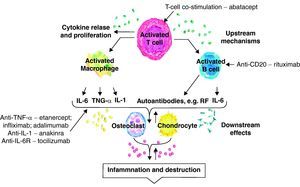
La llegada de las terapias biológicas ha supuesto un gran avance en nuestro arsenal terapéutico al permitirnos el control de los signos y síntomas con un menor deterioro radiológico y proporcionar una mejor calidad de vida a nuestros pacientes. Actualmente disponemos de 8 agentes biológicos para la AR con diferentes dianas de actuación (fig. 1): a)5 contra el factor de necrosis tumoral (anti-TNF) que son el receptor soluble, etanercept (ETN), y 4 anticuerpos monoclonales, infliximab (IFX), golimumab, certolizumab pegilado y adalimumab (ADA); b) un anticuerpo monoclonal contra linfocitos B, rituximab (RTX); c) una proteína de fusión moduladora de la activación de células T, abatacept (ABA); d) un anticuerpo monoclonal contra el receptor de la interleucina 6 (IL-6), tocilizumab (TCZ), y e) un inhibidor de la interleucina 1 (IL-1), anakinra. Un problema con el que nos encontramos en la práctica diaria es la gran variabilidad que presentan los pacientes en respuesta a las diversas terapias biológicas, de tal modo que entre un 20 y un 40% no van a responder1. Lógicamente esto supone un alto coste económico a la vez que va a exponer de forma innecesaria a pacientes no respondedores a efectos adversos. De aquí deriva la importancia de hallar factores predictores tanto de respuesta a estas terapias como de efectos secundarios de las mismas, confiando que en un futuro sea posible la aplicación de una medicina personalizada basada en el genotipo de cada paciente. Sin embargo, en nuestra revisión no hemos encontrado estudios que valoren la existencia de factores que permitan predecir efectos secundarios de los diferentes agentes biológicos.
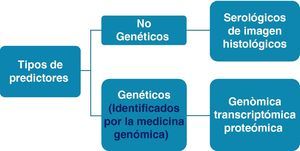
Clasificación de los predictores de respuesta a los agentes biológicosDe una manera sencilla podemos clasificar los factores predictores de respuesta en no genéticos y genéticos (fig. 2); estos últimos identificados por medicina genómica que abarca la genómica, transcriptómica y proteómica. Los no genéticos a su vez se clasifican en clínicos, serológicos, de imagen e histológicos.
Partiendo de una población heterogénea con AR, encontraremos diferencias en la distribución de proteínas y anticuerpos (factor reumatoide [FR], anti-CCP, etc.), ARN (transcripción de genes), ADN (polimorfismos, microsatélites), respuesta a fármacos y manifestaciones clínicas. A partir de esas diferencias podremos definir predictores.
Predictores clínicosAunque existen en la literatura especializada múltiples estudios con respecto a su valor pronóstico en la AR, son escasos los estudios que han estudiado su valor como predictores de respuesta. Posiblemente el más importante haya sido el de Hyrich et al2 que analizan dicha asociación en una cohorte de 3.646 pacientes. Concluyen que la baja discapacidad física al inicio del tratamiento, estimada por HAQ, se comporta como un predictor de respuesta a la terapia anti-TNF mientras que un HAQ basal alto es un fuerte predictor de ausencia a ella. Con respecto al sexo, las mujeres alcanzaron menor porcentaje de remisión, al igual que ocurrió con el hábito tabáquico en relación con la respuesta a IFX. Por su parte ni la edad, ni la duración de la enfermedad ni el DAS 28 previo a la terapia resultaron ser predictores de respuesta. En este mismo estudio destacamos que el uso de metotrexato asociado al tratamiento anti-TNF (ETN o IFX) actuó como un fuerte predictor de respuesta, en tanto que el fallo previo a varios fármacos antirreumáticos modificadores de la enfermedad (FAME) se acompañó de un menor porcentaje de remisión.
Predictores serológicosFactor reumatoideDentro de este tipo de predictores, la positividad del FR y una mejor respuesta a es la asociación más importante descrita. En un trabajo de Hyrich et al4 la presencia del FR se asoció a una menor respuesta a anti-TNFα. Otro estudio5 previo alcanzó la misma conclusión pero en relación con el isotipo IgA, de modo que los pacientes con altos niveles de éste presentaron una pobre respuesta.
Anticuerpos antipéptido citrulinadoHoy en día parece establecido que su presencia en sangre se asocia a mejor respuesta a RTX4. Una «mayor actividad de linfocitos B», cuya expresión sérica sería FR y anti-CCP podría explicar la mejor respuesta a RTX observada en estos pacientes. En contraposición, la positividad de este anticuerpo se relaciona con una menor respuesta a anti-TNF y bajos niveles a mejor respuesta a la terapia combinada IFX+FAME4,6.
CitocinasA pesar de parecer factores predictores de respuesta atractivos, dado que son dianas de las terapias biológicas, ninguno de los estudiados han demostrado de modo concluyente y uniforme ser un factor predictor de respuesta. Existen diversos estudios en esta línea, entre los que destacamos el de Fabre et al7 en el que valores basales altos de proteína quimiotáctica de monocitos 1 (MCP-1), de factor de crecimiento epidérmico (EGF), así como la combinación de este último con valores basales altos de PCR se asociaron a respuesta a ETN a los 3 meses de tratamiento.
Proteína oligomérica de la matriz cartilaginosaLa proteína oligomérica de la matriz cartilaginosa (COMP) es considerada un marcador de recambio del cartílago articular y por tanto podría predecir el daño óseo que acontece en la AR. En este sentido, un estudio8 demuestra que pacientes con valores basales bajos de COMP responden mejor a los 3 meses a ADA, con más de un 50% de ellos alcanzando un ACR70. En cambio, Lequerre et al no encontraron que los niveles basales de esta proteína se asociaran a una respuesta clínica a IFX.
Predictores de imagenTeóricamente se postula que la vascularización sinovial valorada por eco-Doppler previa al tratamiento biológico podría asociarse a variaciones en la respuesta. En nuestra revisión no hemos encontrado estudios que valoren específicamente datos de imagen como predictores de respuesta.
Predictores histológicosAunque disponemos de resultados muy limitados respecto a predictores histológicos de respuesta a biológicos, recientemente Klaasen et al9 identificaron la presencia de agregados linfocíticos sinoviales como un predictor de respuesta a IFX.
Predictores genéticos o identificados por la medicina genómicaLa región genética complejo mayor de histocompatibilidad (HLA) va a explicar aproximadamente un tercio del componente genético de la AR. Sin embargo, el resto del componente genético responsable de la AR se está mostrando muy difícil de identificar. La farmacogenómica va a investigar las variaciones del ADN y ARN relacionadas con la respuesta a un fármaco determinado. Dentro del genoma humano las principales variaciones son: microsatélites (repeticiones de una secuencia corta), minisatélites (repeticiones de una secuencia larga), variaciones en el número de copias (grandes fragmentos de ADN), inserción o deleción (pérdida o adicción de una pequeña secuencia de nucleótidos) y los polimorfismos de un solo nucleótido (SNP). Los SNP son las variaciones más frecuentes del genoma y por ello objeto de múltiples estudios en diversas enfermedades, abarcando lógicamente a pacientes con AR (tabla 1).
Importantes polimorfismos en la artritis reumatoide.
| Gen | Posición del polimorfismo | Alelos | Posible efecto del polimorfismo |
| TNF-α | -238 | G | Erosiones articulares más graves |
| A | Erosiones articulares menos graves | ||
| -308 | G | Producción normal de TNF-α | |
| A | Regulación positiva de la producción de TNF-α | ||
| -857 | C | Puede contribuir a la susceptibilidad a AR | |
| T | Alta producción de TNF-α | ||
| IL-1RN | -1087 | G>A | Desconocido |
| IL-1 | IL-1 α+4845 (exón 5) | G | Producción alterada de IL-1 α. Incremento de la susceptibilidad a AR |
| T | |||
| IL-10 | -1082 | G | Regulación positiva de la producción de IL-10 en los linfocitos |
| A | Concentraciones bajas de IL-10. Se asocia a AR en mujeres | ||
| BAT2 | Microsatélite | Desconocido | |
| HLA | Alelos específicos del epítopo compartido (HLA-DR) | Puede contribuir a la susceptibilidad y gravedad de la AR | |
| TNFRSF1A | -609 | G>T | Desconocido |
| TNFRSF1B | 676 | T>G | Desconocido |
A: adenina; BAT2: transcrito 2 asociado al alelo HLA-B; C: citosina; G: guanina; HLA: antígeno de histocompatibilidad; IL-1: interleucina 1; IL-10: interleucina 10; IL-1RN: antagonista del receptor IL-1; IL-6: interleucina 6; T: timina; TNFRSF: receptor de TNF-α; TNF-α: factor de necrosis tumoral alfa.
La técnica del estudio de asociación de genoma completo (genomewide association o GWAS) ha supuesto un gran avance al permitir analizar cientos de miles de SNP en todo el genoma, mientras que con las técnicas clásicas sólo se estudiaban determinados SNP de una pequeña región del genoma humano11. Los estudios GWAS identifican la distribución de SNP en casos y controles para posteriormente observar si existen diferencias estadísticas en su distribución en ambos grupos. Esto ha permitido identificar SNP de diferentes poblaciones o individuos y con ello polimorfismos que se correlacionan con susceptibilidad a enfermedades o respuesta a fármacos.
Aunque son escasos este tipo de estudios en pacientes con AR, el primero realizado valorando la respuesta a anti-TNF se llevó a cabo en 200812. Entre sus conclusiones destacaron la asociación del SNP2985 con cambios en el DAS28, del gen rs1800896 (alelo G) en el promotor de la IL-10 y del locus de la paraoxonasa (PON1) con una buena respuesta a anti-TNF. La paraoxonasa es una enzima relacionada con lipoproteínas de alta densidad que parece tener un papel importante en la respuesta inflamatoria.
Con respecto a ETN, Padyukow10 en un estudio observacional retrospectivo no encontró asociación entre el epítopo compartido y respuesta. Sin embargo, los pacientes con el genotipo IL-10-1087 G/G y TNF α-308 G/G (sujetos con respuesta inflamatoria baja) presentaron una mejor respuesta.
Otros 3 trabajos13 obtuvieron resultados similares de asociación entre los polimorfismos TNF-α-308G y una mejor respuesta a ETN en comparación con el genotipo -308 A/G.
Entre los estudios que valoran respuesta a IFX, Marotte et al en su estudio prospectivo longitudinal en 198 pacientes no encontraron asociación entre alelos del epítopo compartido y respuesta. Varios grupos14 han coincidido en que el SNP –308 G/A en la región promotora del TNFα influye en la respuesta a IFX, de modo similar a lo comentado respecto a ETN, el genotipo GG determinaría una mejor respuesta a IFX mientras que el AA tendría peor respuesta.
A pesar de todos estos estudios relativamente concordantes, un metaanálisis13 muy reciente que incluye 13 estudios y 1.817 pacientes, concluye que ni el polimorfismo TNFα-308A/G ni los alelos del epítopo compartido se asocian con respuesta a la terapia anti-TNF (ETN, IFX, ADA) y únicamente encuentra asociación entre el polimorfismo TNFα-238A/G y respuesta a IFX (asociación de alelo A con pobre respuesta).
Predictores genéticos identificados por análisis transcriptómicoEl desarrollo de las micromatrices (microarrays) permite estudiar toda la actividad transcripcional de un tipo celular o un tejido. Supone un importante avance dado que previamente sólo era posible estudiar simultáneamente la expresión de un pequeño número de genes.
Mediante esta técnica Julià et al15 analizan la expresión génica de células sanguíneas a partir del ARN de 44 pacientes con AR antes de iniciar IFX y valora la respuesta a la semana 14 según criterios EULAR. Concluyen que 8 genes (HLADRB3, SH2D1B, GNLY, CAMP, SLC2A3, IL2RB, MXD4, TLR4) predicen la respuesta a IFX. En otro estudio16 reciente del mismo grupo se analizó la expresión génica de células sanguíneas, células T CD4 y células B mediante microarrays en 9 pacientes con AR antes de iniciar RTX y valoran la respuesta a la semana 24 (DAS28). La sobreexpresión del gen TRAF1 se asoció a una favorable respuesta mientras que la del gen ARG1 lo hizo con peor respuesta a RTX.
Predictores genéticos identificados por análisis proteómicoLa proteómica será una importante línea de investigación futura para identificar predictores de respuesta a biológicos, aunque por el momento hay pocos estudios publicados y con series pequeñas.
ConclusionesPara poder llevar a cabo una medicina personalizada, que permita seleccionar mejor a los pacientes respondedores y evitar posibles efectos secundarios asociados a estas terapias, se precisa la identificación de factores predictores de respuesta. Estos pueden clasificarse en genéticos y no genéticos. Posiblemente a día de hoy sólo los factores séricos, positividad del FR y anti-CCP permiten predecir la respuesta a determinados agentes biológicos. Sin embargo, se necesitan más estudios para poder definir de modo inequívoco factores predictores de respuesta a las distintas terapias biológicas y descubrir nuevos. Para ello se deberá minimizar la heterogeneidad de los estudios en lo que se refiere a: diseño, tamaño muestral —la mayoría de los estudios genéticos publicados constan de menos de 100 pacientes—, características clínicas basales, criterios de respuesta (EULAR/ACR), tiempo de valoración de la respuesta al tratamiento y tecnología empleada, sobre todo en aquellos identificados por análisis transcriptómico o proteómico.
En un futuro, la generalización de los estudios mediante GWAS y secuenciación completa del genoma mediante técnicas de segunda generación en grandes poblaciones de pacientes con AR nos permitirá probablemente obtener una combinación de múltiples SNP y posiblemente un score para predecir la respuesta a un determinado tratamiento biológico. Es esperanzador avizar en el horizonte que la combinación de estos SNP con diversos predictores no genéticos, en su mayoría identificados mediante transcriptómica y proteómica, nos permitirán ajustar el tratamiento biológico más adecuado a cada paciente.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores de este artículo agradecen a la investigadora Esther Baena Chaparro su apoyo y ayuda en la realización del mismo.