Evaluar la expresión génica del gen IP-10 en pacientes con lupus eritematoso sistémico (LES) y su posible relación con la actividad de la enfermedad.
Pacientes y métodosEl estudio incluyó 120 pacientes diagnosticados con LES y 30 controles sanos. Se investigó la expresión génica relativa de IP-10 con el método fold change, la cual fue correlacionada con el nivel de actividad lúpica evaluado con el instrumento SLEDAI 2-K.
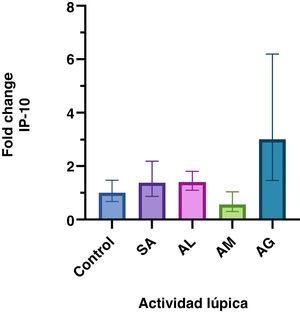
ResultadosSe encontraron diferentes niveles en la expresión génica de IP-10 relacionada con la actividad lúpica (p =<0,001). Estos fueron mayores en los pacientes con actividad grave respecto a aquellos sin actividad, baja y moderada. El incremento en la expresión génica del grupo con actividad grave fue significativo con un fold change de tres.
ConclusiónEl incremento significativo en la expresión génica relativa IP-10 puede ser un marcador de actividad lúpica grave.
To evaluate IP-10 gene expression in patients with SLE, and its possible relationship with disease activity.
Patients and methodsThis study included 120 patients diagnosed with SLE and 30 healthy controls. The relative gene expression of IP-10 was investigated with the Fold Change method, which was correlated with the level of lupus activity evaluated with the SLEDAI 2-K instrument.
ResultsDifferent levels of gene expression were found according to the SLE activity (p =<0.001). IP-10 gene expression levels were higher in patients with severe activity than in those with no activity, low activity, and moderate activity. The increase in gene expression in the severe activity group was significant with a Fold Change of 3.
ConclusionThe significant increase in relative gene expression IP-10 may be a marker of severe lupus activity.
El lupus eritematoso sistémico (LES) es una enfermedad autoinmune crónica, de presentación clínica heterogénea, actividad variable en el tiempo, multisistémica, caracterizada por la presencia de anticuerpos antinucleares1,2.
A pesar de décadas de estudios, pocos biomarcadores de actividad de LES han sido validados3. Los marcadores convencionales (anti-C1q, anti-dsDNA, C3 y C4) son subóptimos, pues se les adjudica una sensibilidad del 50% y una especificidad del 75%4. Es por ello, que se requieren nuevos biomarcadores para conocer la respuesta al tratamiento, así como la necesidad de modificarlo.
Hay evidencia de que la señalización del interferón (IFN) es inductora cardinal en la fisiopatología del LES. Por ejemplo, el uso de INF para otras patologías induce anticuerpos asociados a LES. Por lo tanto, la sobreexpresión de genes estimulados por INF en la sangre de estos pacientes tiene potencial de ser biomarcadora de actividad5.
La quimiocina proteína 10 inducida por INF (IP-10 o CXCL10) es secretada por macrófagos, monocitos y células endoteliales en respuesta al INF6. Su receptor CXCR3 se expresa en linfocitos Th1, B, NK y células dendríticas. Entre sus funciones, se incluyen la actividad quimiotáctica, la inducción de apoptosis, la regulación de crecimiento, la proliferación celular y la angiogénesis. Su importancia radica en que sus niveles séricos han sido vinculados con actividad y nefritis lúpica en diversos estudios7.
Si bien se ha encontrado un aumento en la expresión relativa de IP-10 en los leucocitos obtenidos de sangre periférica de pacientes con LES8, la información que correlacione el grado de expresión génica con la actividad es escasa, es por ello que el objetivo de este trabajo es evaluar la expresión génica relativa del gen IP-10 en personas con LES, y su posible relación con la actividad de la enfermedad.
Pacientes y métodosPacientesDurante el período comprendido entre enero del 2018 a octubre del 2019, se reclutaron 270 mujeres que acudieron al servicio de consulta externa de Reumatología, o bien fueron ingresadas en el Hospital Central Militar, clasificadas como casos de LES, según los criterios de American College of Rheumatology de 1997. Su actividad fue cuantificada mediante el instrumento SLEDAI-2K a ciegas de la obtención de la expresión génica de IP-109, quedando excluidas aquellas con procesos infecciosos activos en los últimos tres meses.
También, se reclutaron 30 mujeres sanas para el grupo control que no tuvieran obesidad, enfermedades autoinmunes o infecciones activas en los últimos tres meses. Todas las pacientes otorgaron su consentimiento informado y esta investigación fue aprobada por el comité de bioética e investigación en el Hospital Central Militar.
Se formaron cinco grupos con base en su puntaje SLEDAI-2K cada uno con n = 30: 1.- control, 2.- sin actividad (SA) cero a dos puntos, 3.- actividad leve (AL) tres a cuatro puntos, 4.- actividad moderada (AM) seis a ocho puntos y 5.- actividad grave (AG) > ocho puntos3.
MuestrasFueron obtenidas muestras de sangre por punción venosa de todas las pacientes, que se procesaron en el Laboratorio de Biología Molecular de la Escuela Militar de Graduados de Sanidad de la SEDENA, obteniéndose y cuantificándose el ácido ribonucleico (ARN) total de los leucocitos en cada una de las muestras.
Además, se diseñaron primers para amplificar una región especifica en el exón 2 y 3 del gen IP-10 y se midió la expresión génica, usando Real-Time Quantitative Reverse Transcription PCR (qRT-PCR) de ARN mensajero (ARNm) de IP-10. Posteriormente, se obtuvo la expresión de un gen endógeno (gliceraldehído-3-fosfato deshidrogenasa [GAPDH]) de todas las muestras. El kit utilizado fue KAPA SYBR FAST One-Step qRT-PCR Master Mix (2X), de la empresa Kappa Biosystem.
Cuantificación de la expresión génicaLa cuantificación de la expresión génica relativa de los grupos se obtuvo mediante el método «fold change» (FC), usando los valores de los ciclos en los cuales las muestras comienzan a presentar fluorescencia significativa (CT), obtenidos mediante qRT-PCR; los valores CT son inversamente proporcionales a la concentración inicial de ARNm.
Para este método, el primer paso es obtener el ΔCT que es la diferencia entre los promedios CT del gen estudiado y del endógeno en un grupo, dicho valor evalúa la expresión del gen en un grupo. Luego, se obtiene el ΔΔCT que es la diferencia de los ΔCT entre los grupos con la enfermedad y el de control, este evalúa el comportamiento del gen en una patología. Finalmente, se obtiene el FC mediante la fórmula 2-ΔΔCT y significa las veces que un gen está sobre o sub-expresado en la escala logarítmica, en un grupo o grupos con LES respecto al grupo control que tiene un valor FC de 1.
Por su escala continua, tendencia a la normalidad y no representar una proporción, la significancia estadística global entre grupos se obtiene con los valores ΔCT mediante el test de análisis de varianza (ANOVA). Se considera que la expresión del gen en estudio (IP-10) está significativamente involucrada en la actividad de la enfermedad cuando el FC del grupo es mayor a dos.
El análisis estadístico se realizó mediante el software GraphPad Prism 8.4.0.
ResultadosLos datos que no tienen distribución normal se presentan como mediana (IQR). De todas las pacientes incluidas, la mediana de edad fue de 38 años (29, 53). Las mujeres con LES tuvieron una mediana SLEDAI-2K de ocho puntos (cuatro, 27) y una edad de 42 años (34, 54). Las características por grupo y el análisis FC se muestran en la tabla 1 y en la figura 1 se grafica el FC de los grupos. La diferencia en la expresión relativa de IP-10 entre grupos fue estadísticamente significativa (F = 5,3 [4.104,8], p =<0,001) determinada por la prueba ANOVA de una vía (Brown-Forsythe).
Características de los grupos y expresión génica relativa (fold change) de IP-10
| No. = 150 | Control | SA | AL | AM | AG | ||
|---|---|---|---|---|---|---|---|
| No. = 30 | No. = 30 | No. = 30 | No. = 30 | No. = 30 | |||
| Características por grupo | p | ||||||
| Edad | Mediana (IQR) | 24(23,28) | 34 (27, 54) | 37 (31,49) | 47 (34, 69) | 43 (34,54) | < 0.001* |
| Edad LES | 34 (27, 54) | 37 (31,49) | 47 (34, 69) | 43 (34,54) | 0,3* | ||
| SLEDAI-2K | 0 (0, 0) | 4 (4, 4) | 7 (6, 8) | 25 (20, 34) | |||
| Análisis fold change de IP-10 | p | ||||||
| GADPH | Media | 17,1 | 17,6 | 17,7 | 17,6 | 17,7 | |
| IP-10 | 24,2 | 24,31 | 24,39 | 25,6 | 23,3 | ||
| ΔCT | 7,1 | 6,7 | 6,6 | 8 | 5,5 | < 0,001** | |
| ΔΔCT | 0 | -0,46 | -0,49 | 0,8 | -1,59 | ||
| Fold change | Media IC 95% | 1 (0,6 - 1,4) | 1,38 (0,8 - 2,1) | 1,41 (1,1 - 1,8) | 0,56 (0,3 - 1) | 3 (1,4 - 6,1) |
AG: Actividad grave; AL: Actividad leve; AM: Actividad moderada; Edad LES: Edad de pacientes con lupus eritematoso sistémico; GAPDH: Gliceraldehído-3-fosfato deshidrogenasa; IC: Intervalo de confianza; IQR: Rango intercuartílico; IP-10: Proteína 10 inducida por interferón; SA: Sin actividad.
Expresión génica relativa de IP-10 de acuerdo con la actividad de LES. Se presenta la media geométrica y el IC 95% del fold change de IP-10 en cada grupo. Solo el grupo con actividad grave presenta un incremento significativo en la expresión génica relativa.
AG: Actividad grave; AL: Actividad leve; AM: Actividad moderada; LES: lupus eritematoso sistémico; SA: Sin actividad.
Este estudio demostró un incremento significativo (FC > 2) en la expresión génica de IP-10 en los pacientes con LES y AG, que presentaron un mayor aumento en la expresión de IP-10 respecto a los otros sujetos. Con excepción del grupo AM, los valores de FC fueron más altos en las personas diagnosticadas con LES.
Si bien, hubo diferencia estadísticamente significativa entre la edad de todos los grupos, cuando se consideró únicamente a los pacientes diagnosticados con LES, el valor p fue de 0,3. Por lo que no se puede considerar que la variación en la expresión génica observada en el presente trabajo esté asociada con la edad.
Los hallazgos en el grupo con AM podrían explicarse por diferencias en la respuesta al tratamiento, ya que se ha encontrado que la expresión IP-10 está disminuida en pacientes con buena respuesta al tratamiento inmunomodulador. Es posible que el grupo con AM represente a la población con una buena respuesta, los pacientes con AL o SA, los que inician terapia o son de reciente diagnóstico y aquellos con AG resistente al tratamiento10.
También hay evidencia de que la predominancia del fenotipo Th1 en linfocitos CD4+ de pacientes con LES se relaciona con una mayor actividad lúpica. El INF secretado por linfocitos Th1 promueve la síntesis y liberación de los productos de respuesta a INF; como IP-10. En cambio, las interleucinas secretadas por los linfocitos Th2 antagonizan la secreción y acción de mediadores liberados por linfocitos Th1; entre ellos INF11. Por consiguiente, la menor expresión génica de IP-10 del grupo con AM puede deberse a un predominio de linfocitos Th2, teniendo el grupo con AG una mayor expresión génica de IP-10 por predominancia de linfocitos Th1.
En conclusión, se puede sugerir que el incremento significativo en la expresión génica IP-10 puede ser un biomarcador de actividad lúpica grave, lo cual implica que podría ser utilizada para identificar aquellos pacientes que requieran un ajuste de tratamiento y una vigilancia más estricta. Al tratarse de un estudio preliminar, no se recogieron los tratamientos de los pacientes ni el fenotipo de los linfocitos CD4+, por lo que nuevos estudios prospectivos con mayor número de casos que consideren estas variables son necesarios para corroborar nuestras observaciones.
FinanciaciónEste trabajo no ha recibido ningún tipo de financiación.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.